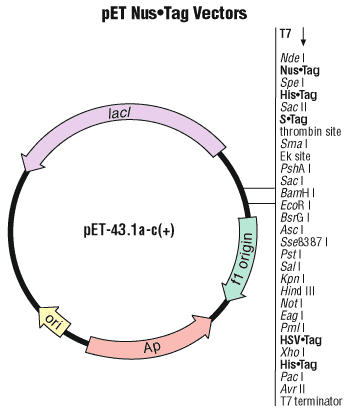
NusA技術標簽載體pET NusA,pET-44 vector (pET-44a(+), pET-44b(+) and pET-44c(+)
- 價 格:¥2930
- 貨 號:BiovectorNusA.
- 產 地:北京
- BioVector NTCC典型培養物保藏中心
- 聯系人:Dr.Xu, Biovector NTCC Inc.
電話:400-800-2947 工作QQ:1843439339 (微信同號)
郵件:Biovector@163.com
手機:18901268599
地址:北京
- 已注冊
| pET NusA Fusion Systems 43.1 and 44 | |||||||||
 | |||||||||
| Production of soluble, active proteins in E. coli The pET NusA Fusion Systems are designed for cloning and high-level expression of polypeptide sequences fused with the 495 aa NusA (Nus?Tag?) protein. The NusA protein has been identified as having the highest potential for solubility when a data base of more than 4000 E. coli proteins was subjected to solubility modeling (1, 2). Greater than 85% of the expressed protein was soluble in tests with each of four different NusA fusion proteins (1). The pET-43.1 and pET-44 vectors incorporate the Nus?Tag fusion partner and are also compatible with the trxB/gor mutant Origami? Origami B, Rosetta-gami?, and Rosetta-gami B strains, which facilitate disulfide bond formation in the cytoplasm (2). Disulfide bonded proteins may be obtained by employing the combination of the pET-43.1 or pET-44 vector and these trxB/gor host strains Features | |||||||||
| 1. Harrison, R.G. (2000) inNovations 11, 4–7. 2. Davis, G.D., Elisee, C., Newham, D.M. and Harrison, R.G. (1998) Biotechnol. Bioeng. 65, 382–388. 3. Novy, R. and Drott, D. (2002) inNovaations14, 12–13. | |||||||||
| Product Description | |||||||||
The pET NusA Fusion System 44 is designed for cloning and high-level expression of polypeptide sequences fused with the 495 aa NusA (Nus?Tag?) protein. The solubility of many target proteins in the E. coli cytoplasm is increased when they are expressed NusA fusions. The pET-44a-c(+) vectors, which are derived from the corresponding pET-43.1 series, encode an additional N-terminal His?Tag? sequence for enhanced yields when purifying a target protein with His?Mag? Agarose Beads from lysates generated with the BugBuster? HT Reagent and PopCulture? Reagent. N-terminal S?Tag? and optional C-terminal HSV?Tag? and His?Tag fusion sequences are also available for versatile detection and purification of target proteins. ?TB055 pET System Manual Components:
|
NusA技術:顯著增強大腸桿菌表達可溶、活性蛋白
前言
NusA融合蛋白表達系統于9年前(1999年)由Davis發明,并在2000年由Novagen首先進行商業化設計和開發,特別推薦用于以NusA作為N端標簽時提高在大腸桿菌中難溶的重組表達蛋白的可溶性。NusA融合蛋白表達系統面市至今,已經通過一些高通量表達篩選的評估確認其在可溶性改善方面的有效性。關于NusA融合表達蛋白去除或不去除NusA標簽,目的蛋白仍然具有活性的報道也有很多。正是由于NusA標簽系統的這種特性,使其成為目前在大腸桿菌表達體系中使用最為廣泛的三個融合標簽之一(Cabrita,2006)。NusA提高蛋白可溶性的機制一直被研究探討,已經獲得一些認識。本文總結和回顧這些關于NusA系統性能的研究報道。
RealTime ready 智力大沖浪!答對5題,即獲贈美國傲仕優質保溫杯!
各種“難溶蛋白”與NusA標簽重組表達顯著提高可溶性
在眾多NusA標簽融合蛋白可溶性研究報告中,有三篇文章因為其研究所涉及的蛋白的數量多和異源性范圍廣而得到研究者的重視。這些研究結果被總結在表1中。其中,Shih等(2002)的研究由于包括了來自四個不同物種的目標蛋白而尤為重要。在Korf等(2005)的研究中,他們發現20℃誘導比30℃誘導的NusA標簽蛋白有更好的可溶性。另外,使用NusA系統驗證,八個較大分子量蛋白(>70kDa)中的七個得到了可溶性表達,而其它的融合標簽(GST,MBP和hexahistidine)系統則只得到了四個可溶性表達的蛋白。
表1.用大量的目標蛋白評估NusA標簽對提高融合蛋白可溶性的作用
參考文獻a | 目的蛋白數目 | 目的插入序列種屬 | 目標蛋白大小范圍 | NusA融合蛋白可溶性比例 |
Shih等(2002) | 40 | 酵母,哺乳動物,植物,昆蟲 | 9-100 | 60 |
Korf等(2005) | 75 | 人 | 6-127 | 60b |
Kohl等(2008) | 96 | 人 | 1-118 | 44c |
a. Korf等和Kohl等的研究中包含了六組氨酸標簽。
b. 可溶性蛋白量大于等于10%即認為該融合蛋白可溶。
c. 純化后的融合蛋白如果在SDS-PAGE后考染在合適位置出現條帶即認為可溶。
Korf等的還發現對于定位于真核細胞細胞器,質膜或者骨架的蛋白,相對于其它標簽系統來講,NusA標簽是最好的可溶性表達的選擇。Kohl等(2008)也發現只要在20-25℃誘導表達,NusA標簽能夠大大提高難表達的蛋白比如膜蛋白的可溶性。與Korf等的研究結果一致,Kohl等也發現25℃誘導表達比30℃或37℃誘導表達可以純化得到更多的NusA融合蛋白。
切除NusA標簽獲得后保持活性且正確折疊的蛋白
表2總結了16個采用NusA標簽成功獲得可溶性蛋白,在切除標簽后這些蛋白仍有正確折疊結構和活性。大部分這種研究是是關于分子量小于或接近20kDa的目標蛋白。純化后的目標蛋白產量范圍在1.5-100mg/L。趨化因子和細胞因子可以得到高達30-100mg/L的產量。其它關于這些蛋白表達和純化的有參考價值信息包括:
■ 植物磷酸烯醇式丙酮酸—羧化酶激酶(Ermolova2003)——目標蛋白切除標簽后用BDA(藍色葡聚糖)親和層析樹脂純化。純化后蛋白的催化活性比未切除標簽的融合蛋白高50倍。
■ Xklp3a,Tep3Ag和E8R(De Marco2004)——用蛋白酶切割后,His-融合的TEV和NusA被Ni2+離子親和色譜選擇性去除。與Ni2+親和結合的標簽被緊密地結合在樹脂上,在流出液中則可以得到純化的目的蛋白。所有這三種蛋白在純化后都正確折疊且均一分散在溶液中。純化的膜結合蛋白E8R牛痘病毒蛋白在Tris緩沖液中除去NusA后出現了沉淀,然而加入0.02%的月桂酰基麥芽糖苷和150mM的氯化鈉后,蛋白又重新變得可溶。
■ 環麥芽糖糊精酶(Turner2005)——這個蛋白屬于α-淀粉酶家族。這個家族的蛋白通常在大腸桿菌中很難以活性形式表達出來。將其與腸激酶混合孵育24小時以上會使其活性逐漸增強,直到達到未經腸激酶處理過的融合蛋白的2倍以上,這也說明標簽的存在降低了該酶的活性。可以用固化了Cu2+的親和層析柱去除切除的融合標簽。
■ 八種人趨化因子(Magis-trelli 2005)——所有的蛋白都在OrigamiTMB菌株中表達提高它們在胞質中的二硫鍵形成率。在趨化因子編碼序列的C端引入了AviTMTag(親和素)生物素化序列。切割后的細胞趨化因子可以用單體的親和素樹脂親和層析與切割下的NusA標簽和蛋白酶混合物分離開。所有切割純化后的蛋白在細胞趨化實驗中都顯示了活性,而沒有一個融合蛋白有這樣的活性。
■ 蚯蚓血紅蛋白(Karlsen2005)——酶切后,用分子篩分離純化蚯蚓血紅蛋白,純化后的蛋白通過圓二色譜檢測得到的α-螺旋結構與模型預期結果一致,且純化后的蛋白可以以單體的形式穩定保存。
■ 人白介素-29(Li2006)——用S-蛋白親和層析比Ni2+親和層析可以得到更純的目的蛋白。將融合蛋白N端的NusA/His?Tag?/S?Tag?標簽切掉后,用鏈親和素瓊脂去除生物素標記的凝血酶。用水皰性口膜炎病毒(VSV)處理固定的人羊膜上皮細胞(WISH細胞)后,通過檢測純化的IL-29對細胞的保護效應來檢測其抗病毒活性。
■ 人干擾素-λ2(Li2007)——酶切后,用Novagen提供的EKaptureTM瓊脂除去重組的腸激酶。先用純化后的干擾素-λ2處理WISH細胞,24小時后加入VSV病毒,可以觀察到干擾素-λ2可以有效地保護細胞免于病毒介導的病變。
表2.切除NusA標簽獲得后保持活性且正確折疊的蛋白
參考文獻 | 目的蛋白 | 目的蛋白分子量(kDa) | 切割用蛋白酶 | 融合蛋白親和層析固定介質 | 純化后目的蛋白產量(mg/L) |
Ermolova等(2003) | 植物磷酸烯醇式丙酮酸羧化酶激酶 | 32 | 凝血酶 | Ni2+ | 1.5 |
DeMarco等(2004) | Xklp3A Tep3Ag E8R | 15 NRa 32b | TEV酶 TEV酶 TEV酶 | Ni2+ | 5.0 2.5 4.0 |
Turner等(2005) | 環麥芽糖糊精酶 | 69 | 腸激酶 | Cu2+ | 1.6 |
Magistrelli等(2005) | 八種人趨化因子 | 8-21 | Xa因子 | Ni2+ | 30-100 |
Karlsen等(2005) | 蚯蚓血紅蛋白 | 15 | TEV酶 | Ni2+ | NRa |
Li和He(2006) | 人白介素-29 | 20 | 凝血酶 | S-蛋白 | 60 |
Li和Huang(2007) | 人干擾素-λ2 | 20 | 腸激酶 | Ni2+ | 65 |
a. 未報道
b. 根據NCBI報道預測的全長蛋白分子量
與NusA標簽融合且具有活性的蛋白
與這些切除NusA標簽后保持活性且正確折疊的蛋白不同,還有很多報道指出目的蛋白在“NusA-目的蛋白”的融合形式時具有很好的活性。比如單鏈(ScFv)催化活性抗體14D9(Zheng2003),來自Aequorea victoria的綠色熒光蛋白(Nallamsetty2006),人二氫葉酸還原酶(Nallamsetty 2006),來自Ensis directus蟶子的精氨酸酶激酶(Compaan2003),來自B. thuringiensis的修飾δ-內毒素(Kumar 2005),人BCMA跨膜受體(Guan2006),植物α-雙加氧酶1(Liu 2006),以及來自Plasmodiumfalciparum的b-ketoacyl-acyl載體蛋白合成酶(Lack2006)等,反映了各種不同背景的蛋白都顯示出了與NusA標簽融合后的活性。
NusA標簽提高蛋白可溶性的可能機制
Houry(1999)等揭示NusA蛋白是分子伴侶GroEL在體內的必須底物。而GroEL與其共作用因子GroES是大腸桿菌唯一的在所有生長條件下必需的分子伴侶系統。Douette等(2005)研究了融合蛋白NusA-UCP1(uncouplingprotein1)的可溶產量。UCP1是一種線粒體膜蛋白。這些作者發現16℃培養時,當GroEL共過表達的情況下,融合蛋白的可溶性有更大的提高。這個結果也表明NusA與分子伴侶途徑相作用,從而阻止參與蛋白的聚集。
總結
已有充分的證據證明NusA標簽系統能顯著提高多種不同來源蛋白的可溶性表達,而這些蛋白在單獨表達時往往形成不可溶的包涵體。在一些研究報告中,用蛋白酶切除NusA標簽能使目的蛋白仍保持正確折疊和生物學活性;相反,在另外許多報道中也指出當目的蛋白與NusA融合而非切除時,融合蛋白也同樣具有活性。NusA標簽系統的成功至少部分地是由于其與大腸桿菌分子伴侶系統相互作用的結果。
參考文獻
Cabrita, L.D., et al. 2006. BMC Biotechnol. 6, 12.
Compaan, D.M. and Ellington, W.R. 2003. J Exp. Biol. 206, 1545.
Davis, G.D., et al. 1999. Biotechnol. Bioeng. 65, 382.
De Marco, V., et al. 2004. Biochem. Biophys. Res. Commun. 322,766.
Douette, P., et al. 2005. Biochem. Biophys. Res. Commun. 333,686.
Ermolova, N.V., et al. 2003. Protein Expr. Purif. 29, 123.
Guan, Z.B., et al. 2006. Sheng Wu Gong Cheng Xue Bao 22, 46.
Houry, W.A., et al. 1999. Nature 402, 147.
Karlsen, O.A., et al. 2005. FEBS J 272, 2428.
Kohl, T., et al. 2008. Proteome Sci. 6, 4.
Korf, U., et al. 2005. Proteomics 5, 3571.
Kumar, S., et al. 2005. J Inverteb. Pathol. 88, 83.
Lack, G., et al. 2006. J Biol. Chem. 281, 9538.
Li, M. and He, S. 2006. J Biotechnol. 122, 334.
Li, M. and Huang, D. 2007. Biotechnol. Lett. 29, 1025.
Liu, W., et al. 2006. Plant Physiol. Biochem. 44, 284.
Magistrelli, G., et al. 2005. Biochem. Biophys. Res. Commun. 334,370.
Nallamsetty, S. and Waugh, D.S. 2006. Prot. Expr. Purif. 45,175.
Shih, Y.P., et al. 2002. Protein Sci. 11, 1714.
Turner, P., et al. 2005. Prot. Expr. Purif. 39, 54.
WilkInson, D.L and Harrison, R.G. 1991. Biotechnol. (N.Y.). 9,443.
Zheng, L., et al. 2003. J Biochem. 133,577.
- 公告/新聞